シングルセルデータを使った代謝活性予測
先週のCellに以下の興味深い論文が出ていたので手法を読み解いていく。この論文では、Flux balance analysisをscRNA-seqデータへ応用することで各細胞の代謝状態を推定するCompassというツールを開発し、Th17のサブセット間で代謝状態が如何に異なるかを推定、評価している。
Flux balance analysis (FBA)
FBAは化学量論 (Stoichiometry) と代謝流量 (Metabolic flux) のみに着目して代謝状態をモデリングする方法。
Stoichiometryとは「Acetyl-CoA + L-Glutamate <=> CoA + N-Acetyl-L-glutamate」のような反応式で表される、代謝反応の入力・出力の量的関係にまつわる情報を指す。
FBAでは対象生物種の全ての代謝反応を同時にモデリングする。上述の論文ではRecon 2というヒト代謝マップのリファレンスデータを使っている。
定式化
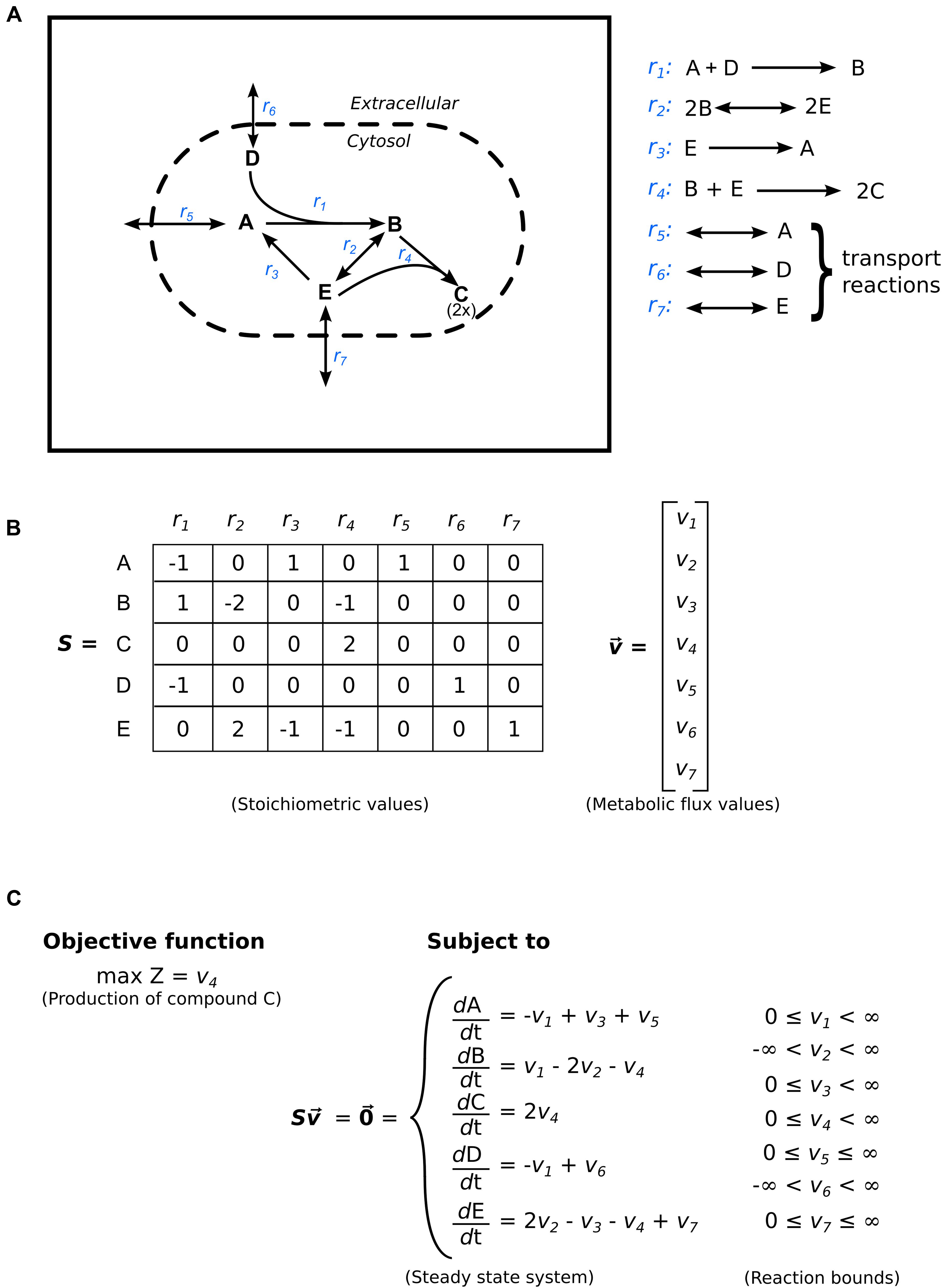
FBAでは全代謝反応のStoichiometryとMetabolic fluxを下図Bのように行列Sとベクトルvで表す。行列Sの行は代謝物、列は代謝反応に対応しており、各行列成分には反応によって産生される物質量が格納される。例えば代謝反応が「A + D → B」という反応であれば、産生される代謝物Bの行に1、消費される代謝物AとDの行に-1が格納される。
FBAでは、対象となる代謝マップを持つ生物において、以下の条件・仮定をおいた場合の「任意の代謝活性の最大値 (または最小値)」を求める。例えば、下図の代謝物Eの産生量 (細胞外への放出量) の最大値を知りたい場合は、下記の条件・仮定下での反応 の流速
の最大値を求める問題を解く。
仮定・条件
引用元:Cuevas et al. “From DNA to FBA: How to build your own genome-scale metabolic model” Front Microbiol. 2016 Jun 17;7:907. doi: 10.3389/fmicb.2016.00907. CC-BY 4.0
上記の条件を満たすは一意には決まらない。これはすなわち対象とする生物種の代謝状態に複数の定常状態があることを意味するが、FBAでは、この複数の定常状態の中から、目的関数が最大・最低になる場合の状態を求める。
線形計画法
これらの目的関数・制約条件は全て1次式なので、FBAでは上記の最適化問題を線形計画法 (Linear programming) を用いて解く。例えば次のような2変数の最適化問題を考える。
- 目的関数
- 制約条件
このとき下図のように問題を捉えることができる。点線で囲まれた斜線部は制約条件を満たす領域である。オレンジの直線はf(x,y)の等高線である。このような領域の中で、ある線形な目的関数が最大・最小となる点はいずれかの頂点であることがわかる。

線形計画法ではこのような目的関数の最大・最小値に該当する頂点を効率よく探索する方法が提案されているのだが、詳しいアルゴリズムに関しては以下の参考文献がわかりやすかった。CompassではCplexというIBMの最適化問題用のPythonライブラリを用いてFBAを実施している。*1
はじめての最適化 | 関口 良行 | 数学 | Kindleストア | Amazon
Compass
ここで肝心の論文に話を戻す。この論文では、上述したFBAと、scRNA-seqでの代謝関連遺伝子の発現情報を利用して、各細胞の代謝状態を推定する方法を提案している。
最大流速  を求める
を求める
最初のステップでは単純なFBAにより各代謝反応の最大流速
を求める。条件(i)は定常状態、(ii)は各代謝反応の可逆性*2・制限*3を表す。ここでは遺伝子発現データは考慮していないため、全ての細胞で
は等しい。

引用元:Wagner et al. “In Silico Modeling of Metabolic State in Single Th17 Cells Reveals Novel Regulators of Inflammation and Autoimmunity” bioRxiv 2020.01.23.912717; doi: https://doi.org/10.1101/2020.01.23.912717 CC-BY 4.0
遺伝子発現を考慮した最適化
遺伝子発現データを元に以下の代謝活性行列とペナルティ行列
を求める。ペナルティ行列の行は代謝反応、列は細胞に対応する。各成分
を以下の通り求める。
ペナルティは代謝に対応する遺伝子が発現が弱いほど1、強いほど0に近づく(論文ではResistance (抵抗)と表現されている)。ここでは各代謝
、各細胞
ごとに
を最小化する以下の最適化問題を解く。(iv)は対象とする代謝rの流速が最大流速
の
倍 *5以上という制約条件なので、代謝r周辺のペナルティ(抵抗)が高いほど
は高い値となる。

引用元:Wagner et al. “In Silico Modeling of Metabolic State in Single Th17 Cells Reveals Novel Regulators of Inflammation and Autoimmunity” bioRxiv 2020.01.23.912717; doi: https://doi.org/10.1101/2020.01.23.912717 CC-BY 4.0
最後にを以下の要領で変換したものをReaction scoreとして解析に利用する。

引用元:Wagner et al. “In Silico Modeling of Metabolic State in Single Th17 Cells Reveals Novel Regulators of Inflammation and Autoimmunity” bioRxiv 2020.01.23.912717; doi: https://doi.org/10.1101/2020.01.23.912717 CC-BY 4.0