問題設計
CellRankは、Fabian TheisとDana Pe'erのグループから最近報告された、シングルセルデータから細胞が将来的に辿る状態遷移の可能性を確率的に求める手法。「細胞Aは細胞Bよりも細胞Cへと辿り着く可能性が高い」といった推定ができる。
Plantir
CellRankのアイデアはDana Pe'erのグループから2019年に発表されたPlantirから発展したものだと思われる。Plantirは、細胞の状態遷移にマルコフ性がある (現在の状態のみが次の状態に影響し、過去の状態は影響しない) と仮定し、上記の課題に挑んでいる。手法の概要は以下の通り*1。
- Diffusion mapでscRNA-seqデータを次元圧縮
- Top nのDiffusion components (DCs) を使いkNN graph
を求める
- 各細胞のPseudo-time
(解析者が選んだ起点からのkNN graphにおける最小パス数) を求める
- Pseudo-timeをもとに
を有向グラフ
に変換する
の場合は細胞
から細胞
へのエッジと考える*2
- 解析者が選んだ終点からのEdgeを
から除外 *3
から細胞間の状態遷移確率行列
を求める
- 状態遷移確率行列
を使ったランダムウォーク
結果として下図dのような結果が得られる。例えば、図dのTrajectoryの(2)に位置する細胞は細胞状態A/B/Cのいずれにも同程度分化できるポテンシャルがあるが、(6)の細胞はB/Cにのみ分化する、といったような解釈ができる。
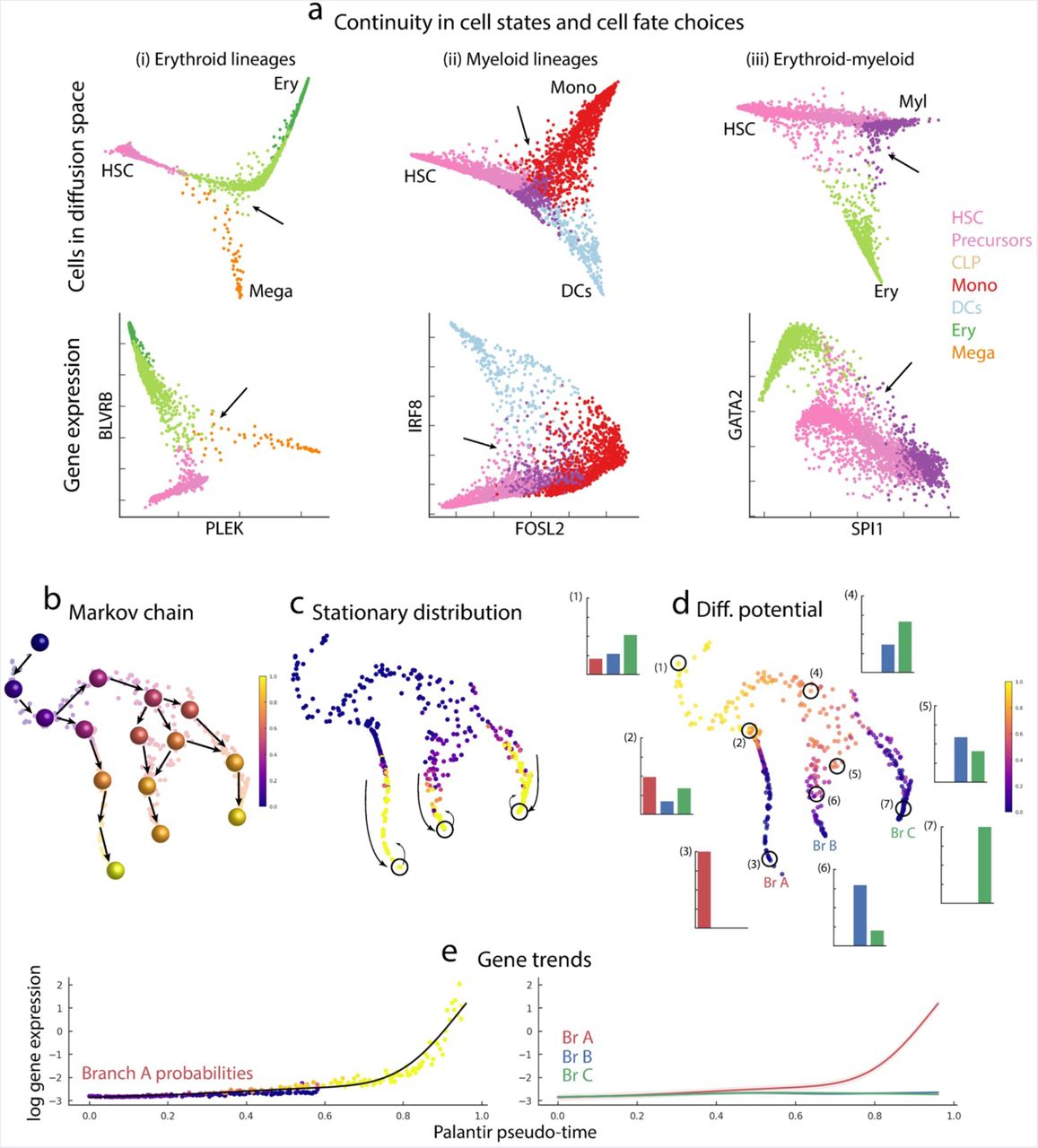
引用元:Setty et al. “Palantir characterizes cell fate continuities in human hematopoiesis” bioRxiv 385328; doi: https://doi.org/10.1101/385328. CC-BY 4.0
CellRank
CellRankはマルコフ連鎖での細胞状態遷移のモデリングにRNA velocity*4を利用する。RNA velocityを使うことによって、(i) 解析者がTrajectoryの始点・終点を決める必要がなく、(ii) Pseudo-timeに依存したPlantirのように「始点から終点へ」という単一方向の状態遷移を仮定する必要がなくなる。したがって、より複雑な細胞状態遷移の過程を調べるのに適した手法だと言える。RNA velocityに関しては以前の記事を参考のこと。手法の概要は以下の通り。7-8のあたりの展開は正直理解できていない。
- kNN graphを作る
- Plantirと違いDiffusion map以外の次元圧縮法を適用可能 (Default: PCA)
- Cell-cell adjacency matrix
- 細胞
と
がいずれかのkNNに含まれていない場合:
- それ以外:
- dist: デフォルトはユークリッド距離
: first top n PCs of cell
(Default: top 30 PCs)
- 細胞
- Connectivity kernel
= row-normalized
- Velocity kernel
- 下図b参照
: 細胞
のVelocity
: 細胞
と
の遺伝子発現の差
: 細胞
のkNN
- VelocityがkNNのどの方向にも向いていない場合に
を小さくする用途のパラメータ
- VelocityがkNNのどの方向にも向いていない場合に
- Merged kernel
- Macrostate identification
- 単一細胞レベルのデータであるカーネル
はノイズが大きいので、細胞集団(Macrostateと呼ぶ)レベルの情報へと変換する
- GPCCA (Generalized Perron Cluster Cluster Analysis)
- 単一細胞レベルのデータであるカーネル
- Macrostate membership
- Macrostate-level transition matrix
- Terminal stateを推定する
- 分化の終末にいるMacrostateは他の細胞への状態遷移が少ないと考えられる。したがってCellRankでは遷移行列
の情報をもとに終末にあるMacrostateを以下のように定める。
- State
is terminal if
: Threshold (Default: 0.96)
- 分化の終末にいるMacrostateは他の細胞への状態遷移が少ないと考えられる。したがってCellRankでは遷移行列
- Initial stateを推定する
- Stationary distribution
を求める
: 定常状態にある細胞状態組成
をMacrostate-levelに変換
- Initial state
- 定常状態を「一定以上の時間が経過した後の細胞状態組成」と仮定すると、Initial stateの細胞は定常状態では少なくなると考えられる。したがってCellRankでは以下の基準で選択する。Initial stateの数
はユーザーが決める必要がある。
- State
is initial if
is in top
smallest values
- 定常状態を「一定以上の時間が経過した後の細胞状態組成」と仮定すると、Initial stateの細胞は定常状態では少なくなると考えられる。したがってCellRankでは以下の基準で選択する。Initial stateの数
- Stationary distribution
- Terminal cells
を定義
- Terminal state
ごとにMembership
の値が高い細胞をTerminal cells
とする
- Terminal state
- 各細胞の運命を推定する
- Transient cellとRecurrent cellという2つの状態を考える。Transient cellからはRecurrent cellへ遷移できるのに対し、反対の遷移は生じないとすると、Transition matrix
は以下のように表せる
- このとき
の
がTransient cell
がRecurrent cellのうち
へ最初に到着する確率を表すことが証明されているらしい*8
- このとき
- CellRankではTerminal cell
をRecurrent cellと見なし、その他の細胞(Transient cell)への遷移をゼロに変換。上述の
を求め、各細胞がどのTerminal stateへ辿り着くか (Absorption probabilityと呼んでいる)を推定する
- Transient cellとRecurrent cellという2つの状態を考える。Transient cellからはRecurrent cellへ遷移できるのに対し、反対の遷移は生じないとすると、Transition matrix

引用元:Lange et al. “ CellRank for directed single-cell fate mapping” bioRxiv 2020.10.19.345983; doi: https://doi.org/10.1101/2020.10.19.345983 CC-BY 4.0
*1:簡略化しているので詳細は論文を要参照
*2:σは
*4:過去記事: https://auroratummy.hatenablog.com/entry/2021/12/07/011122
*5:https://cellrank.readthedocs.io/en/stable/kernels_and_estimators.html#Initialize-a-kernel
*6:https://cellrank.readthedocs.io/en/stable/kernels_and_estimators.html#Compute-a-matrix-decomposition
*7:前述のAdjacency matrixと違うので注意
*8:Tolverらの教科書が引用されていた。http://web.math.ku.dk/noter/filer/stoknoter.pdf