データベース調査③: シングルセルデータベース
HCA Data Portal
Human Cell Atlasで開発されたデータベース。2021年6月時点では55種類のヒト組織に由来する92プロジェクトのシングルセルデータが登録されている。
HCA Data Portalには基本的にユーザーにより登録されたデータが公開されている。Contributeページによると、対象となるシングルセルデータにはいくつかの基準があるようだ。
- Consent: 誰でも自由にアクセス可能なデータが優先
- Sample type: 組織サンプル由来のデータが優先(オルガノイドやCell lineよりも)
- Health status: 健康人由来データが優先
- Organism: ヒト由来データが優先
- Data processing pipeline support: HCAが提供するデータ解析パイプラインに対応しているデータが優先
Exploreページからデータを検索できるが、実際に健康なヒトの組織に由来するデータが多く含まれている印象。メタデータは独自の辞書で標準化しており*1、疾患名や実験条件(e.g. シーケンサー、ライブラリ作製手法、サンプルの保存状態)などで比較的フレキシブルにデータを抽出可能。
ダウンロードできるデータの種類も豊富である。遺伝子発現データ(loom形式)、メタデータ(csv形式)に加えて、生シーケンスデータ(fastq形式)や、HCAのパイプラインで処理されたデータであればマッピングの結果(bam形式)も提供されている。APIでのダウンロードも可能であるらしい。*2
HCAではSmart-seq等の転写産物の全長を読む手法のパイプラインと、Microfluidics/Dropletベースで転写産物の3'端を読む手法(10X Chromium, inDrop, Drop-seq)用のパイプライン(Optimus pipeline)が開発・利用されている。パイプラインはBroad Institute発のワークフロー言語であるWDLで記述されたものが公開されているので、Cromwellなどのワークフローエンジンを使えば誰でもすぐに利用できる。
Single Cell Expression Atlas
EMBL-EBIが開発したデータベース。2021年6月時点では18生物種に由来する217プロジェクトのデータが登録されている。*3
ArrayExpressやGEOに登録されたシングルセルデータを対象としているた。幅広い生物種のデータが登録されている点が特徴。サンプルや細胞のメタデータ(e.g. 生物種名・疾患名、細胞種アノテーション)はEFO(Experimental Factor Ontology)やCell ontologyのターミノロジーを使うことで標準化されている。一方でデータの検索機能はHCA Data Portalの方が充実しており、生物種名やライブラリ作製法で検索できる一方で、疾患や組織名では検索できない。
簡単なデータ可視化が可能であるところは特徴。例えば、"Browse Experiments"から各プロジェクトのページに飛ぶと、どのような細胞サブセットが含まれているかを簡単に調べることができる。

引用元:Papatheodorou et al. “Expression Atlas update: from tissues to single cells” Nucleic Acids Research, Volume 48, Issue D1, 08 January 2020, Pages D77–D83, https://doi.org/10.1093/nar/gkz947. CC-BY 4.0
データは独自のパイプラインで処理されている。HCAと同様にFull-length/3'用にそれぞれ実装されている。パイプラインはNextflowで実装されており、以下のリンクからダウンロード可能。
PanglaoDB
カロリンスカ研究所のグループが開発したデータベース。ヒト・マウス由来のシングルセルデータが登録されている。
PanglaoDBはSRAから取得したシングルセルデータを対象としている。SRAよりダウンロードした生のシーケンスデータに対して独自のパイプラインでデータを処理。他のパイプラインとの違いは細胞種アノテーションを独自開発したツールalonaで自動的に実施するという点。alonaの細胞種アノテーションのアルゴリズムについては以下の論文に記載されている。*4
データ横断的な情報の検索が可能である点が特徴である。Searchページでは、遺伝子名を入力すると、その遺伝子を発現している*5クラスタを全サンプルから探索し、どの細胞種とアノテーションされているクラスタで発現しているかが表示される。

引用元:Oscar et al. "PanglaoDB: a web server for exploration of mouse and human single-cell RNA sequencing data", Database, Volume 2019, 2019, baz046, https://doi.org/10.1093/database/baz046. CC-BY 4.0
Cell type markerページへ飛び、興味のある細胞種の名前を指定すると、彼らが文献調査で収集したマーカー遺伝子と、シングルセルデータを元に計算したマーカー遺伝子の情報にアクセスすることができる。
Ubiquitous index(UI)という指標が計算されている。UIは、PanglaoDBの全クラスタで発現している*6Ubiquitousnessの高い遺伝子が1、ごくわずかなクラスタでしか発現していない遺伝子が0となるような指標である。絶対発現量が高い遺伝子の方がOverestimateされそうではあるが、興味深い。
Single Cell Portal
Broad Instituteで開発されたデータベース。2021年6月現在で、ヒトに限らない様々な生物種に由来する343プロジェクトのデータが公開されている。
HCA Data Portalと目的が被っているような気がするが、もともとはBRAIN (Brain Research through Advancing Innovative Neurotechnologies) initiativeで開発・利用されていたものの、クオリティが高かったからなのか、別のデータにも利用されるようになった、という経緯らしい。
フレキシビリティの高いデータ閲覧が可能であり、tSNE/UMAPなどのEmbeddingに加え、遺伝子名を入力すればViolin plot、Dot plot、Heatmapなどが作成可能である。tSNE/UMAP plot上で特定の細胞を選んでアノテーションをすることも可能。
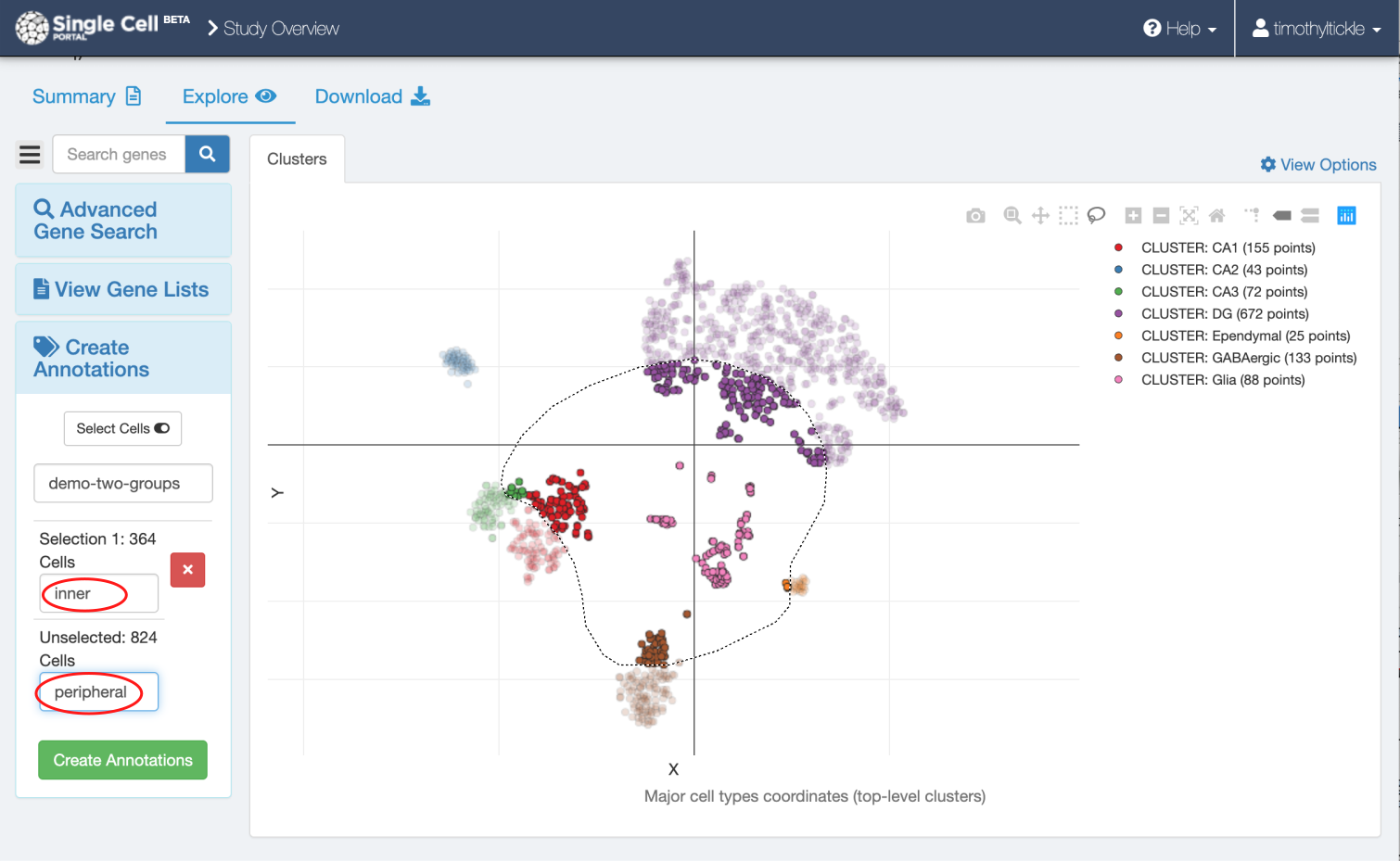
引用元:https://github.com/broadinstitute/single_cell_portal/wiki/Annotations
REST APIも提供されている。
*1:https://data.humancellatlas.org/metadata
*2:https://data.humancellatlas.org/apis マニュアルのリンクが死んでいる
*3:うちヒト由来データは99プロジェクト
*4:そのうち細胞種アノテーションの状況についてもまとめたい
*5:クラスタにおける発現量のMedianが>0であれば発現、という定義らしい
*6:UIの詳細について記述が見つからなかったが、おそらく前述のMedian > 0を「発現あり」とする基準だと思われる