空間オミクスとは
組織中の場所と対応づいたオミクスデータを取得する手法。古くはレーザーマイクロダイセクションで組織断片をくり抜き、そこからオミクスデータを取得するような手間のかかる方法が一般的であったが、最近はより短時間で多くの領域からデータを同時に獲得できるハイスループットな手法が現れている。
空間オミクス解析技術のトップランナーの一人であるJoakim Lundebergのグループから素晴らしいレビューが出ていたのでそれを元に調べてみた。彼らによると空間オミクス解析の手法には大まかに5つのカテゴリーがあるらしい。そのうち以下の3つのカテゴリーに属する手法が汎用性の高いものだと感じた。
- In situ capturing
- In situ hybridization
- In situ sequencing

引用元:Asp et al. “Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration” Bioessays. 2020 Oct;42(10):e1900221. doi: 10.1002/bies.201900221. CC-BY 4.0
主な手法は以下の通り。2013年ごろからたくさんの手法が提案されている中で、Visiumが2019年にリリースされたことで今後利用事例が増えていくものと予想される。また、10X Genomics社は2020年にIn situ sequencingの会社を相次いで買収しており*1、今後はIn situ capturing以外の手法にも参入し、さらに空間オミクス解析へ注力するものと思われる*2。
| 手法 | 引用元 | 解像度 | データ種類 | 販売の有無 | カテゴリー |
|---|---|---|---|---|---|
| Visium | 10X Genomics | 55 μm | Transcriptome | Yes | In situ capturing |
| Visium protein | 10X Genomics | 55 μm | Targeted proteins | Not yet | In situ capturing |
| ST (Original Visium) | Stahl et al. 2015. | 100 μm | Transcriptome | Yes | In situ capturing |
| HDST (Next Visium) | Vickovic et al. 2019. | 2 μm | Transcriptome | No | In situ capturing |
| Slide-seq | Rodriques et al. 2019. | 10 μm | Transcriptome | No | In situ capturing |
| Slide-seq v2 | Stickels et al. 2020. | 10 μm | Transcriptome | No | In situ capturing |
| MERFISH | Chen et al. 2015. | Molecular-level | 100-1000 transcripts | No | In situ hybridization |
| seqFISH+ | Linus Eng et al. 2019. | Molecular-level | 10k transcripts | No | In situ hybridization |
| FISSEQ | Lee et al. 2014. | Molecular-level | 10k transcripts | Yes | In situ sequencing |
| STARmap | Wang et al. 2018 | Molecular-level | 10k transcripts | No | In situ sequencing |
In situ capturing
KTHのJoakim Lundebergらのグループが開発したSpatial transcriptomics (ST)や、Drop-seqの開発者でもあるBroad InstituteのEvan Macoskoらのグループが開発したSlide-seqが本カテゴリーに該当する。10X GenomicsのVisiumはSTを改良した*3ものである。 本手法はポリAを持つオリゴDNAが一定間隔で生えた基板を使う。Permealizationを施した組織切片を貼り付けることで、基板上でmRNAを捉え、逆転写を行うことができる。オリゴDNAには基板上のどの場所から来たかがわかるバーコード配列も含まれており、バーコード情報を元に、配列解読後に同一領域に由来するリードを判別することができる。
手法の特徴は解像度に限界がある一方で、ポリAを持つ転写産物であれば網羅的に検出できることである。Visiumが販売され始めたことから、しばらくはこの手法で得られたデータを最もよく見るようなるものと思われる。

引用元:Asp et al. “Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration” Bioessays. 2020 Oct;42(10):e1900221. doi: 10.1002/bies.201900221. CC-BY 4.0
In situ hybridization
本手法はCaltechのLong Caiらのグループが2014年に発表してseqFISHが発端となり発展しているようだ。seqFISHは蛍光色素をつけたプローブを組織中のRNAに対してハイブリダイズすることを複数ラウンド繰り返す手法。遺伝子ごと&ラウンドごとにプローブにつける蛍光分子を変えることで、RNA分子ごとに蛍光のパターンからどの遺伝子に由来するものであるかを推定することができる。例えば"青 → 赤 → 緑 → 赤"というパターンで検出されたRNAは遺伝子X、といった要領である。
解像度の高さが特徴であるが、測定できる遺伝子の数は限界がある。しかし2019年に発表されたseqFISH+ではプローブのデザインを改良したことで10000遺伝子を同時に計測することに成功しており、改良は進んでいる。プローブを用意するのにものすごく金がかかりそうだが。

引用元:Asp et al. “Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration” Bioessays. 2020 Oct;42(10):e1900221. doi: 10.1002/bies.201900221. CC-BY 4.0
In situ sequencing
本手法は2013年に発表されたPadlock probe / Rolling cycle amplification (RCA) を使う方法が元になり発展しているらしい。Padlock probeは3'端と5'端に結合する領域を持つプローブである。このプローブをハイブリダイズした後に、RCAで増幅、各プローブに固有の領域をSequencing by ligation (SBL)でIn situで配列決定する、という方法。
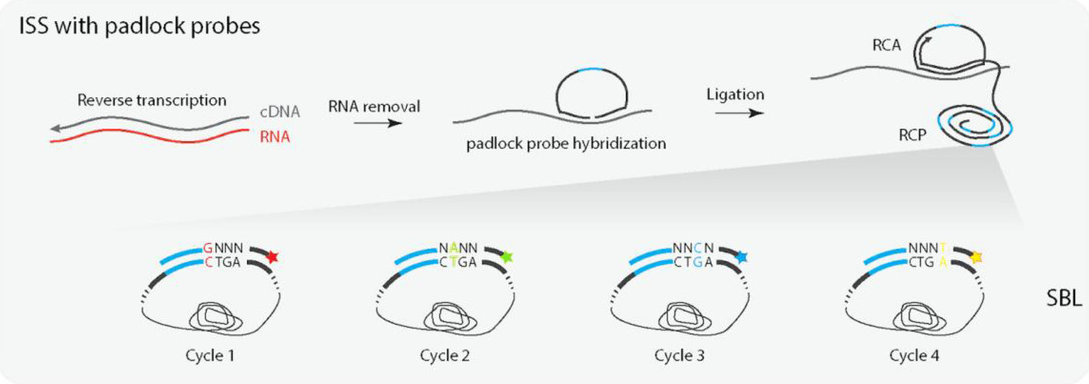
引用元:Asp et al. “Spatially Resolved Transcriptomes—Next Generation Tools for Tissue Exploration” Bioessays. 2020 Oct;42(10):e1900221. doi: 10.1002/bies.201900221. CC-BY 4.0
まとめ
しばらくはVisiumのデータが多く出てくるだろうが、解析手法の覇権争いはまだ終わっておらず、Visium以外のプラットフォームもまだまだ注視する必要がありそう。バイオインフォマティクスではどのような話題があるのだろうか。次はそこを調べたい。
*1:https://www.bio-itworld.com/news/2020/10/05/10x-genomics-announces-two-in-situ-sequencing-acquisitions
*2:株でも買っておこうかしら
*3:解像度が改良されている